クラスター分析
今回はクラスター分析についてまとめる。
クラスター分析はあまりにも有名で、一般向けにも非常にわかりやすい記事がいっぱいある。
クラスター分析は、機械学習の文脈では「教師なし学習」の1つとして紹介されることが多い。
ざっくりとその概要を示すと、何らかの指標によって、個体もしくはある変数を、指標の距離に基づいて、いくつかのグループに分類する方法である。
クラスター分析を行うときには、以下の点を主観的に決定する必要がある。
1. グループ分けの対象
(サンプルを分類するのか、変数を分類するのか)
2. 分類の形式(種類、生成)
(階層的方法 or 非階層的方法)
3. 分類に用いる対象間の距離(類似度)
(ユークリッド距離、マハラノビス距離、コサイン距離 etc)
4. クラスターの合併方法(クラスター間の距離の測定方法)
(ウォード法、群平均法、最短距離法、最長距離法 etc)
5. クラスター数の決定
1については、クラスター分析の目的が決定されれば自動的に決まるので悩む必要はない。というか、社会科学では基本的にサンプルを分類することが多いと思うので、以下ではサンプル(個体)を分類することにしよう。
ただし、2 ~ 4の点は、どれが良いかは一概に言えず悩ましいところである*1。実際に出てきた結果と解釈の適合性との往復で決定していく必要がある。
まず、決定しなければならないのは、2. 分類の形式(種類、生成)である。選択肢は階層的か非階層的かである。
個体が多ければ、階層的クラスター分析はあまり現実的ではない。なぜなら、出力されたデンドログラムを細かく解釈するのがほとんど困難なためである。
階層的クラスター分析では、まず、N個のサンプル数がある場合、それぞれN個のクラスター数があると考える。
このN個のクラスター数を逐次的に「類似」しているものからまとめていき、最終的にN個のサンプルが全て含まれる1つのクラスターにまでしていく過程をデンドログラムを用いて図示する分析である。
なお、ここでの「類似」の指標が、まさに4. クラスターの合併方法(クラスター間の距離の測定方法)で何を指標とするかに関わる。また、基本的にその「類似」は「距離」によって測定されるが、その「距離」にもいくつか種類が存在し、まさに3. 分類に用いる対象間の距離(類似度)に関わる問題である。
ここでは、その方法については特に議論せず、階層的クラスター分析の概要を説明するために先に話を進める。
階層的クラスター分析のアルゴリズムを具体的に表すと、以下のようになる。なお、ここでは簡単のため、サンプルは「スウェーデン、フィンランド、日本、韓国、ドイツ、オーストリア」としよう。
【ステップ1】
全ての個体(クラスター)の中で最も「類似」する個体(クラスター)(例えば、スウェーデンとフィンランド)の組み合わせを同じクラスターとし、新たなクラスターaとする。
【ステップ2】
今、手元にあるのはクラスターa、日本クラスター、韓国クラスター、ドイツクラスター、オーストリアクラスターである。これらのクラスターの中で、【ステップ1】同様に、最も「類似」するクラスター(例えば、日本と韓国)の組み合わせを同じクラスターとし、新たなクラスターbとする。
【ステップ3】
今、手元にあるのはクラスターa、クラスターb、ドイツクラスター、オーストリアクラスターである。これらのクラスターの中で、【ステップ1】【ステップ2】同様に、最も「類似」するクラスター(例えば、ドイツとオーストリア)の組み合わせを同じクラスターとし、新たなクラスターcとする。
【ステップ4】
今、手元にあるのはクラスターa、クラスターb、クラスターcである。これらのクラスターの中で、【ステップ1】【ステップ2】【ステップ3】同様に、最も「類似」するクラスター(例えば、クラスターaとクラスターc)の組み合わせを同じクラスターとし、新たなクラスターdとする。
【ステップ4】
今、手元にあるのはクラスターb、クラスターdである。これらのクラスターの中で、【ステップ1】【ステップ2】【ステップ3】【ステップ4】同様に、最も「類似」するクラスター(例えば、クラスターbとクラスターd)の組み合わせを同じクラスターとし、新たなクラスターeとする。
ここで、手元にあるクラスターはクラスターeの1つとなった。よって、ここでステップを終了する。
以上の流れでは、以下のようなデンドログラムが作成されるだろう。

なお、実際の分析ソフトの出力では縦軸は「非類似度」となる。
このデンドログラムから明らかなように、フィンランド&スウェーデン、オーストリア&ドイツ、日本&韓国 の3つのクラスターとしても理解できるし、フィンランド&スウェーデン&オーストリア&ドイツ、日本&韓国 の2つのクラスターとしても理解可能である。これは、5. クラスター数の決定と関わる。
今回は、最初に全てのサンプルが1つのクラスターを形成していることを前提に述べたが、逆向きのアルゴリズムもある。
すなわち、まず全てのサンプルを含むたった1つのクラスターがあるとして、最も異質性(「類似」していない)の高いクラスターを分岐していく方法である。上記のアルゴリズムの逆verである。
以上が、階層的クラスター分析の概要である。
次に、非階層的クラスター分析を説明する。
非階層的クラスター分析では、まず5. クラスター数を決定する必要がある。ここでは、k個としよう。
【ステップ1】
その後、「中心」となるサンプルをk個ランダムに選ぶ。なお、ここでランダムに選ばれる「中心」となるサンプルによっては、毎回結果が異なることがある。これは、非階層的クラスター分析における初期値依存性の問題である。よって、何回か繰り返しながら分析結果を比較検討する必要がある。特に、初期値が外れ値を選んだ場合、結果が大きく異なることがある。
【ステップ2】
全てのサンプルとk個の「中心」サンプルとの「距離」を測定し、各サンプルを最も近い「中心」と同じクラスターに分割する。したがって、ここではk個のクラスターができたことになる。なお、ここで「距離」の測定の仕方はいくつか開発されており、まさに3. 分類に用いる対象間の距離(類似度)と関わる問題である。ここでは、それについては詳しく解説せず、先に進む。
【ステップ3】
次に、できたk個のクラスターの重心点を求めて、それを新たな「中心」とする。なお、この新しい「中心」、すなわちk個のクラスターの重心点は、もともとランダムに選ばれた最初の「中心」サンプルから移動している点に留意されたい。
【ステップ4】
【ステップ2】に戻る
このステップを繰り返す。最終的には、新たな「中心」ともともとの「中心」が移動しなくなる。その時にできたクラスターが、非階層的なクラスター分析の出力となる。
以上を実際にRでやってみた。
データはirisデータを用いる。irisデータはあまりにも有名だが、念の為以下に補足しておく。
irisデータには5つの変数が用意されていて、それらは「Sepal.Length(ガクの長さ)」「Sepal.Width(ガクの幅)」「Petal.Length(花弁の長さ)」「Petal.Width(花弁の幅)」「Species(種)」となっている。

「Species」以外、量的変数。「Species」には3つの名義変数がある。それらは「setosa」「versicolor」「virginica」である。
なお、サンプル数は150である。
「Sepal.Length」「Sepal.Width」「Petal.Length」「Petal.Width」に対してクラスター分析をかける。なお、クラスター数は3に設定し、「距離」や「類似」の指標についての細かいところは変更せず、とりあえずパッケージのデフォルトで行う。
その後、クラスター分析による分類と「Species」を比べてみる。「Species」によって「Sepal.Length」「Sepal.Width」「Petal.Length」「Petal.Width」は異なると考えられるので、うまく分類されたら、「Species」による分類と類似すると予想できる。
まず、階層的クラスター分析から。

結果は、上の図のようになった。
これによって、3のクラスターができた。このクラスターの分類と「Species」による分類との関係はどのようになっているのだろうか。それをまとめたのが、下図である。

クラスターCは完全に「Species」による分類と一致している。すなわち、クラスターCに分類された個体はsetosaとなっている。
クラスターBはほぼ完全に「Species」による分類と一致している。すなわち、クラスターBに分類された個体はおおよそversicolorとなる。ただし、1つだけvirginicaが混じっている。
クラスターAはversicolorとvirgicaが混在していることがわかる。
それでは、これらのクラスターに分類された個体はどのような個体なのか。
以下に図示。

花のことはよく分からないので解釈に困るが、
クラスターAは「Sepal.Length」や「Petal.Length」が特に長く、全体的に大きめの花。
クラスターBは全体的に中くらいの大きさの花。
クラスターCは「Petal.Length」や「Petal.Width」が極端に短いのが特徴的な花。また、「Sepal.Length」と「Sepal.Width」以外、互いの変数がそこまで相関していないのが特徴的。
次に非階層的クラスター分析
当然だが、階層的クラスター分析のようなデンドログラムは出力されない。
よって、いきなり非階層的クラスター分析による分類と「Species」による分類を比較してみる。結果は、下の図の通り。

クラスター3は完全に「Species」による分類と一致している。すなわち、クラスター3に分類された個体はsetosaとなっている。
クラスター1はほぼ完全に「Species」による分類と一致している。すなわち、クラスター1に分類された個体はおおよそvirginicaとなる。2つだけversicolorが混じっている。
クラスター2はversicolorとvirgicaが混在していることがわかる。
次に、これらのクラスターに分類された個体はどのような個体なのかを以下に図示。
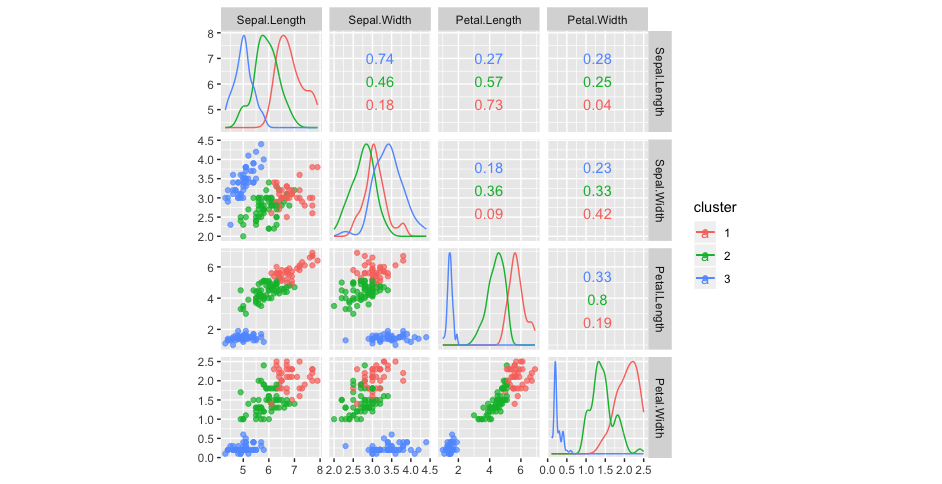
クラスター1は全体的に大きい傾向にある。
クラスター2は全体的に小さい傾向にある。
クラスター3は真ん中くらいか。
互いの相関はこれといった特徴は見受けられれない。
以上である。
なお、上記のRのコードは以下の通り。
#####階層的クラスター分析#####
library(tidyverse)
iris_tibble <- as.tibble(iris) #元のirisデータをtibbleにする
cluster_result <-
iris_tibble %>%
select(Sepal.Length, Sepal.Width, Petal.Length, Petal.Width) %>% #変数取り出し
dist() %>% #距離を計算(defaultはユークリッド距離)
hclust() #階層的クラスター分析実行(defaultは最遠距離法(=完全連結法))
#結果の図示
cluster_result %>%
plot()
#グループ数決定
cluster_result <-
cluster_result %>%
rect.hclust(k = 3)
#ID変数 と クラスタリングされたgroup名変数の結果データを作成
groupA <- rep("A",length(cluster_result[[1]]))
groupB <- rep("B",length(cluster_result[[2]]))
groupC <- rep("C",length(cluster_result[[3]]))
rec <- tibble(
group = c(groupA, groupB, groupC),
ID = c(cluster_result[[1]], cluster_result[[2]], cluster_result[[3]])
)
#irisデータと結果データをIDでリンクする
result <-
iris_tibble %>%
mutate(
ID = 1:length(Sepal.Length) #irisデータにID変数を付与
) %>%
left_join(rec, by = "ID") #ID変数をkeyにデータをmerge
#グループ別にscatter plotの出力
result %>%
ggscatmat(columns = 1:4, color = "group", alpha = 0.7)
#結果テーブル出力
table(result$Species, result$group)
####階層的クラスター分析#####
library(tidyverse)
library(GGally)
iris_tibble <- as.tibble(iris) #元のirisデータをtibbleにする
#非階層的クラスター分析実行
cluster_result <-
iris_tibble %>%
select(Sepal.Length, Sepal.Width, Petal.Length, Petal.Width) %>% #変数取り出し
kmeans(centers = 3) #クラスター数は3
#クラスター分析結果をデータに付与
result <-
iris_tibble %>%
mutate(
cluster = as.character(cluster_result$cluster)
)
#グループ別にscatter plotの出力
result %>%
ggscatmat(columns = 1:4, color = "cluster", alpha = 0.7)
#結果テーブル出力
table(result$Species, result$cluster)
*1:ただし、それを決める指標はいくつか開発されている。