尤度比検定における統計量がカイ二乗分布に従うかの実験
最尤法を用いてパラメータを推定する分析において、モデル1と、モデル1に独立変数を加えたモデル2を比較したい時に便利な検定がある。尤度比検定(likelihood ratio test: LR test)である。最尤法で最大尤度を導出して推定する分析(例えば、ロジスティック回帰分析)なら何でも使えるので非常に便利である。
ただし、モデル1とモデル2がネストされている状態でないと、この検定は使用できない。例えば、y~x1+x2というモデルとy~x1+x2+x3というモデルは尤度比検定で検定可能であるが、y~x1+x2というモデルとy~x3+x4というモデルは比較できない。このように、モデルがネストされていない場合は、情報基準量(AICやBICなど)でモデルの「良さ」を考える。AIC(Akaike Information Criterion)とBIC(Bayesian information criterion)が小さい方が、「良い」モデルとされる。
今回は、情報基準量の話はおいておいて、尤度比検定について考える。
何らかの分析で、ネスト構造にある2つのモデルを回した時に尤度比検定を行うとしよう。この時、以下で定義される統計量が、両者のモデルの最大尤度に差がないという仮説のもとでカイ二乗分布に漸近的に従うとされる。なお、その自由度はモデル1とモデル2の独立変数の差(正確には、推定すべきパラメータの差)である。
今回は、本当に上記の式で定義された統計量がカイ二乗分布に従うのかを実験する。
手順は以下の通り。
①従属変数y(1 or 0)の乱数を1000個発生
②独立変数x1 ~ x5までの乱数を平均50、標準偏差10の正規分布から1000個発生
③model1: y~x1、model2: y~x1+x2、 model3: y~x1+x2+x3、model4: y~x1+x2+x3+x4、model5: y~x1+x2+x3+x4+x5 でそれぞれ二項ロジスティック回帰分析を行う
④上記の統計量をmodel1 vs model2、model1 vs model3、model1 vs model4、model1 vs model5で求める。なお、これらの統計量はそれぞれ自由度1〜自由度4のカイ二乗分布に従うと仮定できる
以上の手続きを1000回繰り返して、そのヒストグラムを確認する。
その後、自由度1〜自由度4のカイ二乗分布から乱数を1000個発生させて、ヒストグラムを確認する。
最後に、両者のヒストグラムが視覚的にどの程度類似しているのかを確認する。
結果は、以下の図の通り。
青色が実験の結果できたヒストグラム、赤色がカイ二乗分布からできたヒストグラムである。
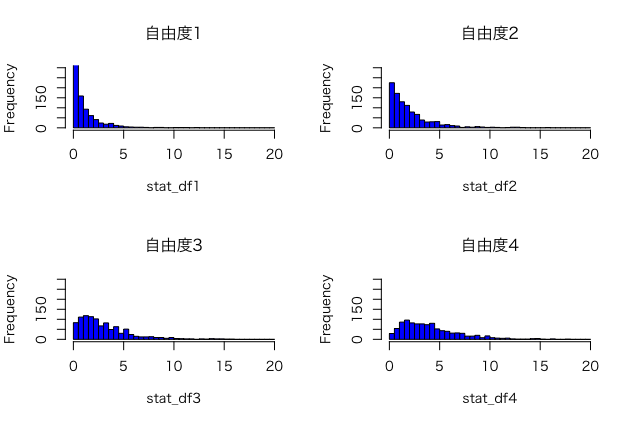

両者とも非常に類似していることがわかる。
以上から、上記の統計量はカイ二乗分布に従うことがわかった。
そのコードは以下の通り。(そろそろ、for()やなんども同じコードを書くのをやめて、もう少しカッコ良いコードを書きたい…。)
N<-1000
n<-1000
stat_df1<-numeric(N)
stat_df2<-numeric(N)
stat_df3<-numeric(N)
stat_df4<-numeric(N)
for(i in 1:N){
y<-round(runif(n,0,1))
x1<-rnorm(n,mean=50,sd=10)
x2<-rnorm(n,mean=50,sd=10)
x3<-rnorm(n,mean=50,sd=10)
x4<-rnorm(n,mean=50,sd=10)
x5<-rnorm(n,mean=50,sd=10)
model1<-summary(glm(y~x1, family="binomial"))
model2<-glm(y~x1+x2, family="binomial")
model3<-glm(y~x1+x2+x3, family="binomial")
model4<-glm(y~x1+x2+x3+x4, family="binomial")
model5<-glm(y~x1+x2+x3+x4+x5, family="binomial")
dev_model1<-model1$deviance
dev_model2<-model2$deviance
dev_model3<-model3$deviance
dev_model4<-model4$deviance
dev_model5<-model5$deviance
stat_df1[i]<-dev_model1 - dev_model2
stat_df2[i]<-dev_model1 - dev_model3
stat_df3[i]<-dev_model1 - dev_model4
stat_df4[i]<-dev_model1 - dev_model5
}
par(mfrow=c(2,2))
par(family="HiraKakuProN-W3")
hist(stat_df1, breaks=seq(0,20,0.5), ylim=c(0, 300), main="自由度1", col="blue")
hist(stat_df2, breaks=seq(0,20,0.5), ylim=c(0, 300), main="自由度2", col="blue")
hist(stat_df3, breaks=seq(0,20,0.5), ylim=c(0, 300), main="自由度3", col="blue")
hist(stat_df4, breaks=seq(0,20,0.5), ylim=c(0, 300), main="自由度4", col="blue")
hist(rchisq(N,df=1), breaks=seq(0,20,0.5), ylim=c(0, 300), main="自由度1", col="red")
hist(rchisq(N,df=2), breaks=seq(0,20,0.5), ylim=c(0, 300), main="自由度2", col="red")
hist(rchisq(N,df=3), breaks=seq(0,20,0.5), ylim=c(0, 300), main="自由度3", col="red")
hist(rchisq(N,df=4), breaks=seq(0,20,0.5), ylim=c(0, 300), main="自由度4", col="red")